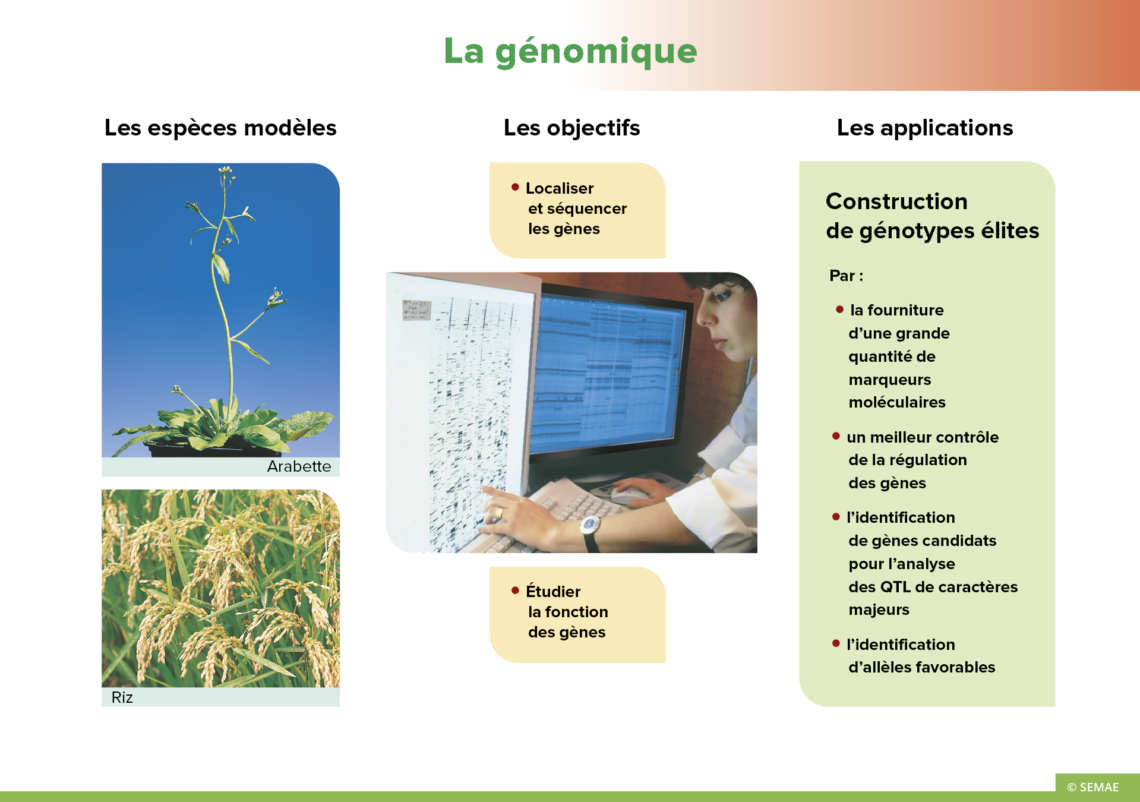
La génomique
La nécessité de disposer de cartes de gènes et l’apparition des cartes à haute densité de marqueurs moléculaires (comme les cartes à puce) ont fait naître des travaux de séquençage systématique du génome. On dispose ainsi d’une représentation physique des chromosomes avec le positionnement des marqueurs et des gènes.
Le terme génomique regroupe les analyses qui consistent à localiser, isoler et séquencer les gènes, puis à étudier leur fonction. Signalons que cette approche ne concerne pas uniquement les végétaux.
Les espèces modèles
L’Arabette des Dames ou Arabidopsis thaliana a été choisie comme espèce modèle pour les programmes de séquençage, car elle cumule un certain nombre de caractéristiques : un génome de taille réduite (100 à 130 mégabases), un cycle de développement rapide (2 mois), une reproduction autogame. Sur cette espèce, de nombreuses techniques sont facilement mises en œuvre comme la transgénèse. De plus, la structure de son génome est conservée dans les espèces apparentées. Les découvertes seront donc directement transposables sur le colza, notamment.
Le riz est également la plante modèle des graminées cultivées, car c’est la graminée cultivée au plus petit génome. De plus, elle possède une bonne synténie avec l’ensemble des autres espèces de ce genre.
Chez le maïs, il s’agit là d’un programme de grande ampleur car la taille du génome est plus importante. On estime que le nombre de gènes est environ 5 fois supérieur à celui d’Arabidopsis.
Le catalogue des gènes
Pour pouvoir accéder à la connaissance du génome des plantes modèles, un inventaire exhaustif des gènes a débuté selon deux approches :
- La première approche est basée sur l’inventaire des gènes exprimés. Les séquences caractérisées sont nommées EST (Expressed Sequence Tags). Toutefois, cette approche a ses limites. En effet, un gène n’est pas toujours actif dans une cellule. Chez Arabidopsis, on estime que 50% seulement des gènes ont pu être répertoriés par cette méthode.
- La seconde approche s’intéresse au séquençage direct du génome. Ce travail est plus long, mais peut donner la séquence complète des gènes et leurs positions.
Vers la construction de génotypes élites
La génomique devrait permettre d’accroître les connaissances dans le domaine végétal et donc d’identifier de nombreux gènes de plantes. Ainsi, les applications sont dans quatre domaines :
- La fourniture d’une grande quantité de marqueurs moléculaires liés à des fonctions ou à des phénotypes. Ceci permet la réalisation de sélection assistée par marqueurs pour des caractères de qualité ou de valeur agronomique.
- Un meilleur contrôle de la régulation des gènes.
- L’identification d’un réservoir de gènes candidats pour l’analyse des QTLs de caractères agronomiques majeurs.
- L’identification d’allèles favorables.
Pour en savoir plus
La génomique est l’étude de l’ensemble du génome d’un organisme, c’est-à-dire le déchiffrage de son ADN et la compréhension de son fonctionnement. Le matériel génétique est généralement organisé en chromosomes, eux-mêmes organisés en paires dans le cas d’organismes diploïdes : il y a alors deux allèles, c’est-à-dire deux formes de chaque gène (une de chaque parent).
Comparés aux génomes animaux, les génomes des plantes sont souvent plus grands mais très variables en taille : 130 Mb pour Arabidopsis, 200 Mb pour le marronnier, 400 Mb pour le riz, 2,4 Gb pour le maïs, 16 Gb pour le blé…
Seule une petite portion du génome code pour des protéines. L’ADN est transcrit en ARNm lui-même traduit en protéines.
La majeure partie de l’ADN présente des séquences dont le rôle n’est pas clairement compris, mais qui font l’objet de recherches intenses (The ENCODE Project Consortium, 2012).
Séquencer un génome puis déterminer son fonctionnement permet de connaître :
- Le nombre de gènes,
- L’organisation du génome,
- L’identification des régions qui ont été affectées par la sélection génétique,
- L’évolution du génome,
- Le rôle des gènes,
- L’influence de facteurs externes comme les facteurs environnementaux sur l’expression du génome.
CARACTÉRISATION DU GÉNOME PAR LES MARQUEURS
Les marqueurs génétiques renseignent sur le génotype d’un individu et ne sont pas modifiés par l’environnement. Ils peuvent être utilisés tout au long d’une expérimentation et sont observables à n’importe quel stade de développement de la plante et sur n’importe quel organe (l’information génétique de la plante est contenue en totalité dans toutes les cellules). Les principaux types de marqueurs génétiques utilisés sont :
- Les marqueurs biochimiques (protéine, isozyme). Les protéines d’une cellule végétale peuvent facilement être extraites et analysées. Les marqueurs biochimiques les plus utilisés sont les isozymes. Ils correspondent aux différentes formes d’une même enzyme et permettent de déterminer la présence de l’allèle correspondant à chacune de ces formes. Dans ce sens, ce sont des révélateurs du polymorphisme entre individus pour les séquences codantes du génome.
- Les marqueurs moléculaires sont des séquences d’ADN aisément détectables. Ils sont utilisés comme balises pour étudier le polymorphisme de l’ADN d’un organisme : substitutions nucléotidiques pour un locus donné ou d’insertion-délétion de segments plus ou moins longs. Ils ne correspondent pas à des gènes actifs mais à des séquences associées aux gènes actifs.
Applications
Grâce aux marqueurs génétiques, il devient possible :
- D’établir l’empreinte génétique d’un individu, c’est à dire de décrire et définir des individus et des variétés en vue de leur inscription, de leur protection et de leur classification,
- De mettre en évidence et suivre les gènes impliqués dans l’expression de caractères d’intérêt agronomique ou technologique.
Caractéristiques d’un marqueur génétique
Un bon marqueur doit être :
- neutre : ses différents allèles n’ont pas d’effet sur le phénotype de l’individu,
- polymorphe : possédant de nombreux allèles permettant de caractériser les différents individus,
- codominant : l’individu hétérozygote peut être distingué car il présente simultanément les caractères de ses parents homozygotes, insensible au milieu, non épistatique, multiallélique.
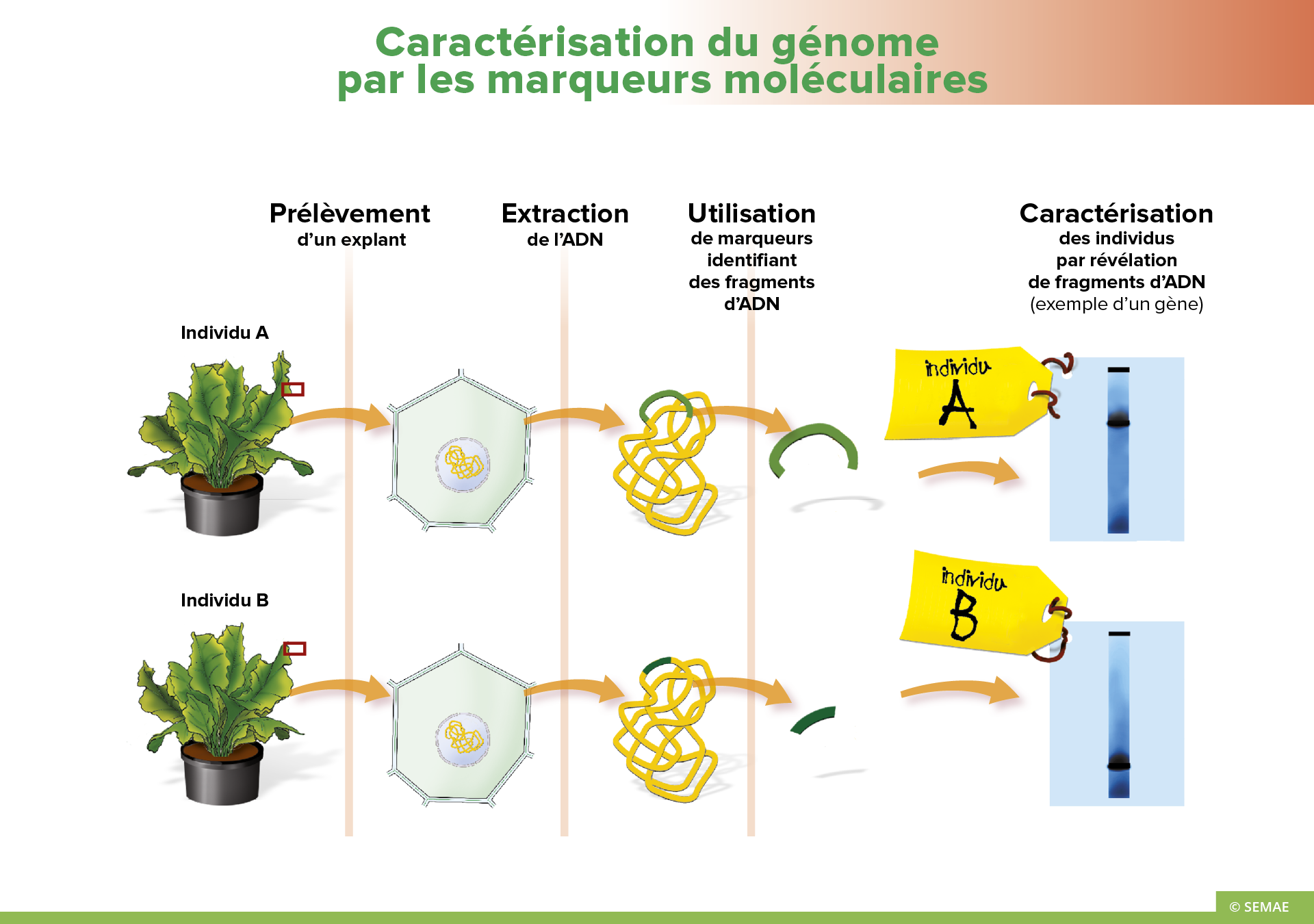
L'EMPREINTE GÉNÉTIQUE D'UNE PLANTE
Les marqueurs moléculaires permettent d’établir l’empreinte génétique d’une plante.
Les puces à ADN permettent de visualiser et de mesurer très rapidement des milliers de différences d’expression au niveau d’un seul nucléotide entre de très nombreux gènes.
Applications
Cette identification peut intervenir à différents niveaux :
- L’identification variétale. Par ces techniques, il devient possible de distinguer les lignées et de reconnaître les parents d’hybrides.
- Le contrôle de la pureté variétale. Il s’agit de détecter des impuretés variétales de lots de semences, ou de tester l’homogénéité d’une population à n’importe quel stade d’un schéma de sélection.
- La protection variétale. Ceci fait partie intégrante de l’identification variétale. Il devient possible de détecter les contrefaçons ou copies de génotypes. Ainsi, pour des questions de droits liés aux obtentions végétales, il a été introduit la notion de variété essentiellement dérivée (VED) : c’est une variété dérivée d’une variété originale par modification de quelques zones chromosomiques réduites.

LES OUTILS COMPLÉMENTAIRES DE LA GÉNOMIQUE
Grâce à la miniaturisation et à l’automatisation des outils de la génomique, le coût et le temps nécessaires à la caractérisation des fragments d’ADN ou de génomes sont en forte diminution d’année en année. Mais pour obtenir des connaissances biologiques à partir des données de génomique, il est nécessaire de réaliser des analyses informatiques complexes ainsi que de très nombreuses mesures et observations sur les plantes. Des laboratoires spécialisés appelés plateformes peuvent prendre en charge ces différents aspects.
Les plateformes bio-informatiques
- Elles intègrent les données produites par les appareils de séquençage,
- Elles assurent l’analyse informatique et statistique des données en produisant des cartes génomiques ou des bases de données et elles guident la recherche vers les gènes actifs ou de régulation les plus intéressants.
Les plateformes de phénotypage
Le phénotypage est l’observation et la mesure du phénotype, c’est-à-dire du caractère d’intérêt. Pour étudier les gènes qui contrôlent un caractère, par exemple la tolérance au stress hydrique, il est nécessaire de mesurer très précisément ce caractère sur de nombreuses plantes grâce à une collection de mutants. En inactivant un gène et en observant les conséquences physiologiques, il est possible de corréler un gène et sa fonction. Les plateformes de phénotypage permettent de réaliser ces mesures à haut débit sur des centaines de plantes différentes, grâce à des robots qui mesurent, pèsent, et photographient les plantes automatiquement et en font des analyses d’image.
Les plateformes de transcriptomique, de protéomique et de métabolomique
Les technologies « omiques » sont des techniques à haut débit qui permettent de générer de grandes quantités de données à des niveaux biologiques multiples : du séquençage des gènes à l’expression des protéines et des structures métaboliques. Ces données peuvent couvrir tous les mécanismes impliqués dans les variations qui se produisent dans les cellules et qui influent la vie de l’organisme.
Elles comprennent la transcriptomique (expression des gènes et de leur régulation), (voir chapitre Les marqueurs moléculaires), la protéomique (étude des protéines), la métabolomique (étude des métabolites cellulaires produits lors des réactions cellulaires) ou encore l’épigénomique (étude des régulations, parfois héritables, de l’expression des gènes qui ne s’accompagnent pas de changement de séquence de l’ADN, via la méthylation ou autres modifications chimiques, ou les ARN non codants).
Des études scientifiques sont déjà parues sur Arabidopsis, plante modèle de laboratoire mais aussi sur les céréales, la pomme de terre ou la tomate. Elles peuvent compléter les études réalisées pour évaluer l’équivalence en substance entre deux variétés.

